Ecco quella proposta da Ernst Mayr in Storia del pensiero biologico (Bollati Boringhieri) tuttora in uso:
Una specie è una comunità riproduttiva di popolazioni, riproduttivamente isolata dalle altre, che in natura occupa una nicchia specifica,
Ricapitolo.
Denny è figlia di una Neandertal e di un Denisovano; il figlio di una balena Peponocephala electra e di un delfino Steno bredanensis è stato avvistato al largo delle Hawaii; e su Nature EcoEvo oggi Axel Barlow et al. scrivono che pure gli orsi bruni Ursus arctos delle foreste attuali hanno ereditato geni da quelli delle caverne, l’Ursis spelaeus estinto da 25 mila anni.
Come la mettiamo?
*
“La CO2 è IL cibo per le piante”
dicono quelli del riscaldamento globale fa solo bene. Su Nature Climate Change, Matthew Smith e Samuel Myers di Harvard simulano con un modello comprendente i 225 alimenti più diffusi, l’effetto di 550 ppm di CO2 atmosferica sui livelli di ferro e zinco disponibile nella dieta degli abitanti di 151 paesi. Stando alle proiezioni demografiche per il 2050, altri 175 milioni di persone soffriranno di carenze di zinco, e 122 milioni di carenze di proteine in media mondiale.
Regions at highest risk—South and Southeast Asia, Africa, and the Middle East—require extra precautions to sustain an already tenuous advance towards improved public health.
Attualmente 1,4 miliardi di donne fertili e di bambini sotto i cinque anni vivono in paesi con un 20% di anemia, nel 2050 saranno di più e nella dieta avranno un 4% di ferro in meno.
La CO2 danneggia prima le donne e i bambini. Com. stampa della Harvard School of Public Health
*
Scienze sociali, crisi delle
In un paper su quattro pubblicati da Nature e sette su diciassette pubblicati da Science fra il 2005 e il 2015, i risultati degli esperimenti non sono riproducibili. E per quelli riproducibili gli effetti sono meno della metà di quelli riferiti…
Ok, il campione è piccolo, 21 in tutto, ma è difficile convincere gli autori a partecipare alle replicazioni e qualcuno disposto a dare soldi e tempo per replicare ricerche in oncologia uscite sulle riviste che più contano, figurarsi quelle in scienze sociali senza neppure una BigPharma dietro… E poi dove si pubblicano i nuovi risultati?
Complimenti a Nature Human Behaviour di settembre, quasi interamente dedicato al lavoro ingrato fatto da Colin Camerer, Brian Nosek e altri volontari del Center for Open Science e ai commenti ingrati degli autori degli esperimenti originali. Camerer et al. avevano anche chiesto a 400 psicologi ed economisti di scommettere soldi virtuali, un po’ come in borsa, su quali risultati sarebbero stati replicati. Hanno scommesso in 217:
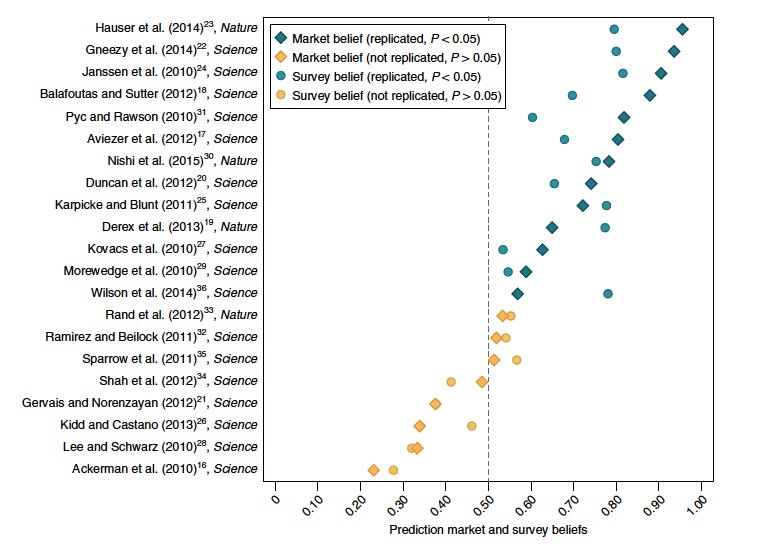
Avevano già capito tutto! Brian Nosek fa un riassunto su twitter e segnala anche le “riforme” in corso.
***
L’ESA ha pubblicato l’immagine composita delle “impronte” radar del lago marziano. Le zone blu, dove la probabilità è maggiore, sono sparse su una ventina di chilometri e potrebbero indicare sia acqua liquida che “sedimenti carichi d’acqua”.
Come la mettiamo con la definizione del grande Mayr?
Come sapevamo già è la migliore definizione operativa per le specie viventi, di cui si possono studiare le popolazioni ma non è una definizione di validità assoluta (d’altro canto non ne esistono). E non solo per i casi di ibridazione come quelli citati, ma soprattutto perchè non si può applicare agli esseri viventi che non hanno riproduzione sessuata.
Per il passato poi, quando le specie sono vicine tra di loro , l’ individuazione di nuove specie da frammenti di singoli individui dei quali non si può conoscere la rappresentatività delle popolazioni di appartenenza , piuttosto che di sottospecie o variabilità interspecifica, ha comunque una base di arbitrarietà. Basti pensare al continuo dibattito su quella o quella specie o non-specie che vede contrapporsi da decenni gli antropologi senza possibilità di verifiche oggettive.
Farei un’ipotesi sulla definizione di specie, anche perchè la domanda è interessante: una specie codificata dal dna, o rna, cellulare che abbia piccola distanza di Hamming fra la codifica dei nucleotidi; sia il fenotipo, che il genotipo, rappresentano qualsiasi carattere della specie, la capacità di riprodursi, e le caratteristiche dell’essere vivente, ed esiste la possibilità di distinguere specie batteriche, e virus, anche in assenza di meiosi.
Quindi avendo due specie di esseri viventi tipo1 e tipo2, ed una classificazione a dna (o rna), una terza specie può essere più vicina ad un genotipo, misurando la distanza di Hamming, e la distanza minore potrebbe caratterizzare l’appartenenza alla specie.
Ok, ho visto che esiste una classificazione degli organismi viventi in base alla distanza genetica
https://it.wikipedia.org/wiki/Distanza_genetica
che permette di costruire lo schema tassonomico, il cladogramma, con particolari distanze genetiche.
Alberto e domenico,
la definizione di Mayr è tuttora quella dei libri di testo, e quelle alternative creano gli stessi problemi- e nessuna è adatta a Denny, geneticamente equidistante da H. neanderthalis e H. denisova, per esempio.
Come scriveva De Queiroz (secondo link)
Definitions of the species category that identify a particular contingent property of metapopulation lineages (including intrinsic reproductive isolation) as a necessary property of species reduce the number of metapopulation lineages that are to be recognized taxonomically as species, but they cause conflicts among alternative species definitions and compromise the status of the species as a basic category of biological organization.
La mia domanda era personale. Con dei colleghi ci chiedevamo se usare “specie” tra virgolette. Magari sì, ma in radio come faccio?
Penso che in qualsiasi tassonomia nota, ci sono individui alla frontiera della specie, che con qualsiasi tipo di metrica (o misura di appartenenza a specie distinte) possono essere considerati appartenenti a due, o più, specie: cioè potrebbe essere esistito un ibrido di H. neanderthalis e H. denisova non classificabile, oppure un ibrido fra più di due specie (più probabile per specie batteriche) non classificabile in una particolare tassonomia.
E’ interessante chiedersi come rappresentare segni non fonetici, come virgolette, sottolineature o evidenziazione, in una rappresentazione teatrale di un testo, oppure in radio: sembra che il linguaggio scritto non sia perfettamente rappresentato da quello parlato, e viceversa.
Penso che esistano alcune possibilità per risolvere il problema, come mettere enfasi nel testo nella lettura, oppure utilizzare un suono di fondo guida standard per la frase, ma servirebbe un rumorista che segua il testo, o l’attore dovrebbe utilizzare pulsanti di attivazione dei suoni di fondo: in fondo le virgolette, o le sottolineature, cambiano i fonemi, come i rumori cambiano i fonemi.
Non vedo perchè usare le virgolette per un termine come quello di specie che sì non è definibile in maniera rigorosa ma è fondamentale nella biologia, come ben spiegato dal Edward Wilson nel suo “La diversità della vita”. Dove si dichiara un sostenitore proprio della definizione di Mayr, pur con i suoi limiti, e spiega perchè considera sbagliato abbandonarla.
Certo Wilson insiste che questa è la definizione di “specie biologica” che assume rilevanza soprattutto per le popolazioni esistenti che si riproducono sessualmente e difficilmente si ibridano in natura con altre specie. Quando si considera invece quella che lui chiama “specie cronologica” , ossia le popolazioni e le loro modifiche su base genetica nel corso dei millenni, sottolinea che ogni specie in fondo è un’ entità transitoria (almeno su tempi lunghissimi). Potrebbe essere utile quindi ricordarlo anche a noi unici Homo sopravvissuti:
oggi siamo ben distinguibili come specie da tutte le altre, anche quelle filogeneticamente più vicine, ma solo poche decine di migliaia di anni fa i nostri antenati facevano tranquillamente sesso con i membri di altre specie “sorelle”, tanto che in ognuno di noi “vivono” i geni non solo del ramo principale ma pure, seppure in minor numero,di quelli collaterali.
In questi giorni continua a ronzarmi in testa una definizione genetica di specie: definendo una specie come l’insieme ordinato dei geni nella sequenza del dna (o rna per i virus), e definendo una specie distinta perchè contenente un numero differente di geni, o una posizione differente dei geni (permutazione), allora per dna con geni distinti la meiosi non è possibile; quindi specie distinte non permettono la riproduzione a causa della diversa lunghezza del dna, o a causa della assenza di sovrapposizione dei geni che porta alla mancanza della codifica di molecole essenziali per la vita (come una malattia genetica nell’embrione a causa della diversità nel dna).
In genere dovrebbe esistere un meccanismo per creare due geni, da un gene, tramite evoluzione (mutazione puntiforme, sostituzione di basi ed altro), così che la trasformazione delle specie dovrebbe essere continua nel tempo, ed individui al confine di specie dovrebbero esistere come trasformazione puntiforme da un gene a due geni.
alberto,
un termine come quello di specie che sì non è definibile in maniera rigorosa ma è fondamentale nella biologia.
La definizione non serve solo ai biologi per capirsi tra di loro. Per es. in USA il coywolf o woyote rientra nella quota cacciabile dei C. lupus (pochi, in pochi stati e soltanto fuori dalle riserve) o dei C. latrans? Nella UE, la direttiva che tutela le cornacchie – e molti altri uccelli – si applica anche agli ibridi di Corvus cornix e di C. corone?
La situazione non migliora con le sottospecie. L’orso grolare è a rischio come i grizzly (divieto totale di caccia, in Canada) o vulnerabile come l’orso polare? Perché gli allevatori della UE sono risarciti per le pecore uccise da un Canis lupus ma non da un ibrido di C. lupus e C. lupus familiaris?
domenico,
sarebbe bello se il processo fosse lineare, vero? Se in una popolazione tutti gli individui avessero lo stesso identico genoma, se durante la vita di ciascuno il Dna non mutasse mai, se non ci fossero tante copie diverse tra loro di tanti geni, se le mutazioni avessero sempre un effetto positivo o negativo invece di essere in maggioranza neutrali, se…
Pensavo che l’idea fosse di distinguere le specie con il genotipo, l’insieme dei geni del DNA o RNA, lasciando libere le modifiche all’interno dei geni, che è usuale nella popolazione a causa delle mutazioni genetiche: quindi i geni come caratteristica diagnostica di una specie, con alcuni geni più importanti per la classificazione, perché alcuni geni sono comuni fra specie.
Quindi, se un gene non cambia molto, la specie è la stessa, se il cambiamento è radicale, allora il gene stesso viene classificato in modo diverso, in quanto le proteine codificate sono diverse, e la specie è diversa.
Pensavo alla tassonomia batterica: se il batterio è semplice, allora le caratteristiche dell’organismo coincidono con l’espressione dei geni; per gli organismi superiori non dovrebbe essere diverso: la tassonomia utilizza i caratteri diagnostici che sono espressioni genetiche.
domenico,
alcuni geni sono comuni fra specie
dipende da che cosa si conta. Per dire, noi, i bonobo e gli scimpanzé abbiamo circa 20-22 mila geni e circa il 98,7% del loro Dna in comune. Se si contano tutte le copie di ogni gene, siccome ne abbiamo di più la differenza aumenta.
Quando molti geni sono diversi, tra eucarioti l’ibridazione diventa impossibile, certo. Ma un gene codifica per decine di proteine, geni identici hanno funzioni diverse negli umani e nei moscerini per esempio, alcune proteine sono espresse da tanti geni ecc. La distanza tra due specie è una stima, non una misura precisa.
Da quanto ho capito, per la tassonomia dei batteri è ancora peggio. Anche se non hanno una riproduzione sessuale, molti sono promiscui e si scambiamo geni creando una confusione pazzesca. Così le specie e le sottospecie continuano a essere rivedute e corrette.
Come le sigle dei geni d’altronde. Uno dei miei preferiti è il gene Shh che s’è scoperto nel moscerino della frutta. Si scrive SHH, HHG1, HLP3, HPE3, MCOPCB5, SMMCI, TPT, TPTPS, sonic hedgehog, Sonic hedgehog, ShhNC – altra confusione pazzesca…
.
Oca
sembra che lei abbia un’ attrazione particolare per gli ibridi; d’ accordo che fanno anch’ essi parte della realtà biologica, ma in termini generali rappresentano un fenomeno quantitativamente limitato. Se poi i burocrati li snobbano per non pagare maggiori indennizzi o complicare le norme, a me non pare una questione interessante.
@Domenico: è la realtà mutevole della variabilità individuale, sia in termini fenotipici (individuata già con grande attenzione da Darwin ma non passata alla cultura collettiva) che in termini di genotipo (non solo in termini di differenti alleli distribuiti nel pool genetico di una popolazione ma pure in diversità di natura diversa come la ripetizione di parti del DNA non codificante o la dislocazione di segmenti o la forma stessa dei cromosomi ed altro ancora) a rendere problematica una ipotetica definizione genetica di specie
alberto,
un’ attrazione particolare per gli ibridi
forse mi confonde con qualcun altro? Ne avrò scritto un paio di volte nella vita.
un fenomeno quantitativamente limitato
penso di no, l’ibridazione interspecifica è un meccanismo dell’evoluzione delle piante sopratutto, ma anche degli animali poliploidi.
Ok, in conclusione, interessante il gene Shh, ma il nome distinto non descrive differenti forme della stesso gene, una codifica standard dovrebbe risolvere il problema (o la definizione del gene con il sequenziamento delle basi dovrebbe risolvere il problema); ma anche la distinzione letterale non cambia la funzione del gene (e la struttura chimica che possiede), come nella nomenclatura chimica delle sostanze.
Per gli organismi diploidi, o poliploidi, sono d’accordo che c’è un problema profondo, è come se una specie possedesse caratteristiche combinate di più specie, e il carattere dominante o recessivo dei geni permette di definire il fenotipo della specie.
La soluzione più semplice è utilizzare ogni espressione genica dell’allele come identificazione di una specie (per esempio nel gruppo sanguigno i geni A, B, 0) e misurare la distanza minima fra combinazioni dei geni (AA, BB, 00, AB, A0, B0) e l’allele della specie con un individuo da testare; ma in questo caso è necessario un sequenziamento della popolazione alla caccia dei geni presenti in modo statisticamente rilevante.
Siccome i geni presenti in organismi simili sono comuni a più specie, allora un database dei geni rilevanti nella tassonomia potrebbe essere utilizzato, anche tenendo conto che le lunghezze dei geni sono equivalenti (database per lunghezza e funzione), anche perché penso che la lunghezza dei geni negli alleli sia equivalente.
Penso che si potrebbe utilizzare un DNA di riferimento con tutte le combinazioni possibili dei geni degli alleli come individuo standard della specie, e misurare la distanza minima di un individuo da testare con tutti i differenti geni negli alleli.
Grazie per questo articolo, e per le interessanti idee.
domenico,
i geni Shh sono stati sequenziati nei vari organismi e se guarda le sigle, vedrà che rappresentano la classifica standard.
si potrebbe utilizzare un DNA di riferimento con tutte le combinazioni possibili
è il primo passo, spesso sotto forma di DNA barcoding, ma con tutte le combinazioni possibili non servirebbe a nulla.
I codici a barre sono nei database (gene libraries, per esempio quella della moscerina della frutta) e nelle riviste escono di continuo metodi matematici/statistici per estrarne informazioni utili.
—
alberto,
combinazione, oggi su Pikaia è uscito un bell’articolo sull’ibridazione/speciazione dei pesci ciclidi.
Oca
il caso della poliploidia è ben noto e relativamente diffuso nelle piante (al contrario che negli animali) ed infatti il Mallet riporta che tra le piante vascolari il 2-7% delle specie ha avuto questa origine. Solo che bisogna ricordarsi che le specie poliploidi sono riproduttivamente isolate dalle specie genitrici diploidi.
Riguardo alla speciazione per ibridazione omoploide, nell’ articolo si parla del fatto che queste specie “COULD be abundant … but nonetheless there are A FEW convincing cases” (maiuscolo mio). Questa scarsità può dipendere dalla difficoltà nel dimostrare che è l’ ibridazione ad aver condotto alla speciazione e magari le analisi genomiche future cambieranno la situazione. Solo che ad oggi i dati verificati indicano qualche decina di specie di ibridi omoploidi (compresi i ciclidi tanto amati da Lorenz) rispetto a molte centinaia di migliaia (con una stima del numero di specie non ancora scoperte che varia tra i milioni e le decine di milioni) di specie complessive esistenti.
I numeri attuali parlano da soli, come pure, quanto ricordato dal Mallet, ossia che a fronte di un 10% circa di specie animali e 25% di piante che sono capaci di produrre individui ibridi con altre specie a livello di popolazioni “interspecific hybrids are of course unusual forming <0,1% of individuals in a typical population”.